大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
DNA甲基化是一种重要的表观遗传修饰,其在基因表达调控、基因组稳定性和细胞分化中起着关键作用。UHRF1(ubiquitin-like, containing PHD and RING finger domains, 1)是一种多结构域蛋白,可作为表观遗传修饰因子发挥作用,能够招募DNMT1以维持DNA甲基化。在DNA复制过程中,UHRF1通过识别半甲基化的DNA和结合组蛋白H3来定位到新合成的DNA上,并通过其E3泛素连接酶活性泛素化组蛋白H3,从而促进DNMT1的招募和DNA甲基化的维持。然而,UHRF1乙酰化修饰及其对DNA甲基化维持的具体作用机制尚不清楚。
近日,广东省人民医院心血管研究所/广东省人民医院医学研究所/香港大学生物医学院王霖晟博士等为第一作者,广东省人民医院医学研究所/香港大学生物医学院/香港大学深圳医院骨科中心周中军教授、广东省人民医院医学研究所金国祥研究员为共同通讯作者,在《Cell Reports》期刊发表题为《MOF-mediated acetylation of UHRF1 enhances UHRF1 E3 ligase activity to facilitate DNA methylation maintenance》的研究论文。研究揭示了MOF(males absent on the first)对UHRF1蛋白的乙酰化修饰如何调节DNA甲基化维持过程。研究发现,MOF能够乙酰化UHRF1的K670位点,而HDAC1(histone deacetylase 1)则能够去乙酰化该位点。这种乙酰化修饰增强了UHRF1的E3泛素连接酶活性,进而促进了组蛋白H3泛素化,这对于DNMT1(DNA methyltransferase 1)的招募和DNA甲基化维持至关重要。深圳易基因在本研究中提供重要的甲基化检测(RRBS)技术服务。

标题:MOF-mediated acetylation of UHRF1 enhances UHRF1 E3 ligase activity to facilitate DNA methylation maintenance(MOF介导的UHRF1乙酰化增强了UHRF1 E3连接酶活性以促进DNA甲基化维持)
期刊:Cell Reports
影响因子:IF 7.5/Q1
技术平台:RRBS等(易基因金牌技术)
本研究结果表明,MOF能够乙酰化UHRF1的K670位点,该位点位于pre-RING连接区,而HDAC1则能够去乙酰化UHRF1的同一位点。此外,当K670发生突变时,MOF还可以乙酰化K667和K668位点。MOF/HDAC1介导的UHRF1乙酰化受细胞周期调控,并在G1/S期达到峰值,这与UHRF1招募DNMT1以维持DNA甲基化的功能相一致。此外,UHRF1乙酰化显著增强了其E3泛素连接酶活性。在这些位点上消除UHRF1乙酰化会减弱UHRF1介导的H3泛素化,进而影响DNMT1的招募和DNA甲基化。综合来看,这些发现确定了MOF是UHRF1的乙酰转移酶,并明确了通过MOF介导的UHRF1乙酰化调节DNA甲基化维持的机制。
研究亮点
- MOF在K670位点乙酰化UHRF1,这一过程可被HDAC1消除。
- MOF介导的乙酰化增强了UHRF1的E3泛素连接酶活性。
- 消除UHRF1乙酰化会抑制组蛋白H3泛素化及DNMT1招募。
- UHRF1的K670位点乙酰化为DNA甲基化维持的正常功能所必需。
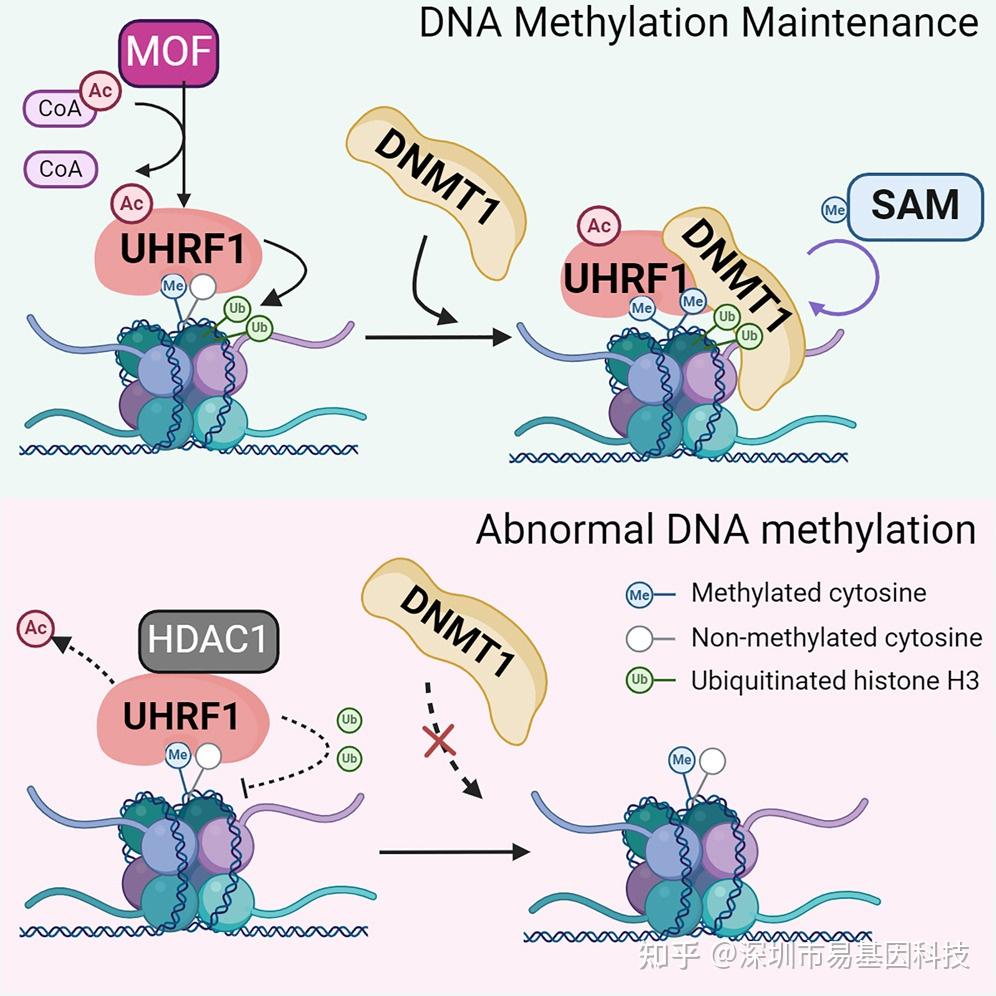
研究摘要
研究方法
细胞实验:在HEK293T细胞中进行了一系列细胞实验,包括共转染不同组的质粒、免疫共沉淀、免疫荧光染色等,以研究UHRF1乙酰化修饰及其对DNA甲基化的影响。
体外实验:通过体外乙酰化实验,使用纯化的GST-UHRF1和GST-MOF蛋白,验证了MOF对UHRF1的直接乙酰化作用。
质谱分析:利用质谱技术分析了MOF介导的UHRF1乙酰化位点,确定K670为主要乙酰化位点,同时K667和K668也可以被乙酰化。
基因敲除和敲低实验:通过siRNA敲低MOF或在小鼠胚胎干细胞(ESCs)中敲除UHRF1,研究其对DNA甲基化的影响。
DNA甲基化分析:采用免疫荧光染色、简化基因组亚硫酸盐测序(RRBS)等方法,分析DNA甲基化水平和模式的变化。
结果图形
(1)MOF和HDAC1分别是UHRF1的乙酰转移酶和去乙酰化酶
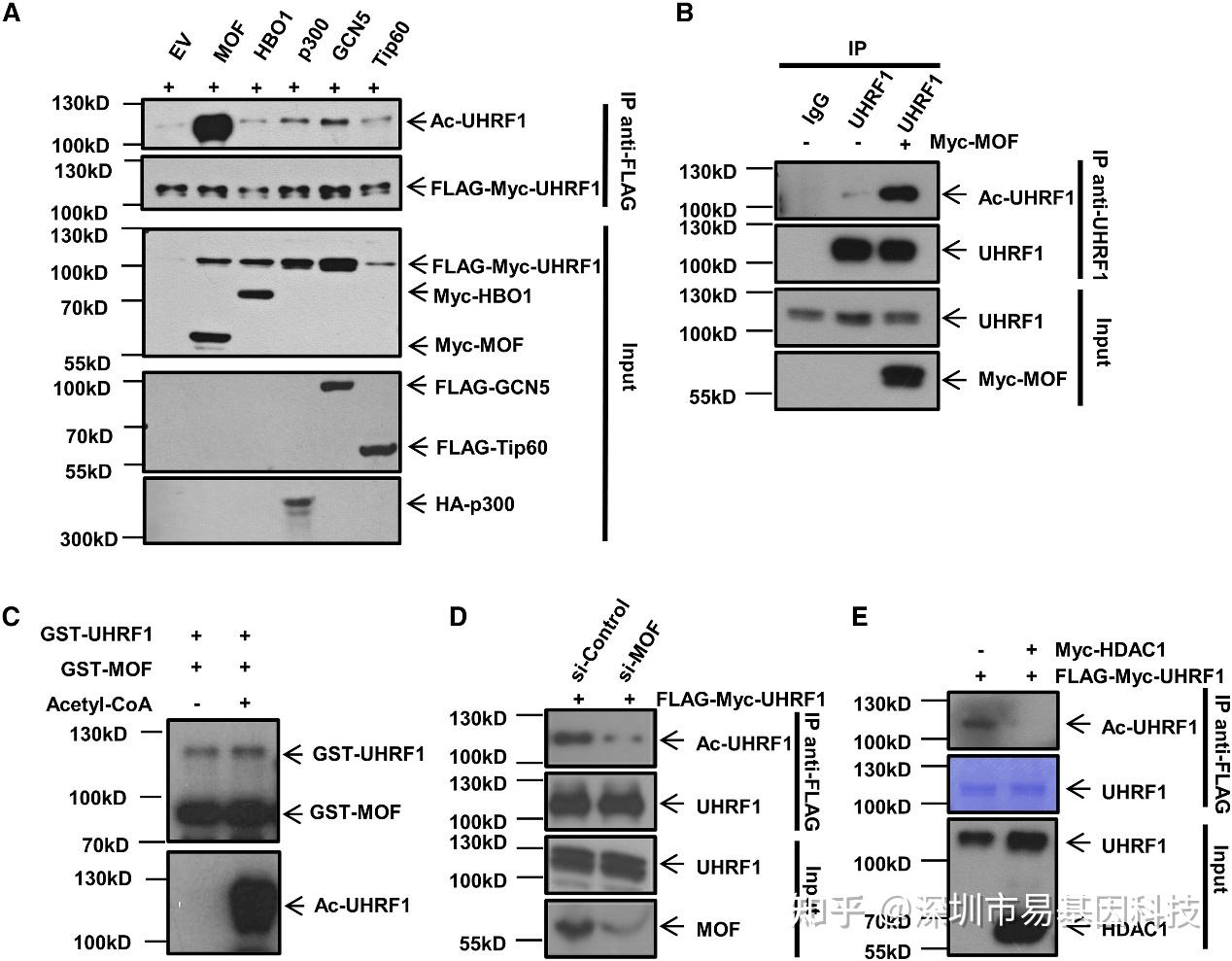
图1:UHRF1特异性地被MOF乙酰化,并被HDAC1去乙酰化。
(A)在HEK293T细胞中共表达FLAG-Myc-UHRF1和不同的组蛋白乙酰转移酶(HATs)。使用FLAG抗体对细胞裂解物进行免疫沉淀,并通过乙酰化赖氨酸抗体进行免疫印迹分析以检测乙酰化的UHRF1。EV代表对照组。
(B)将空载体或Myc-MOF质粒转染到HEK293T细胞中。转染48小时后,对内源性UHRF1进行免疫沉淀,并使用UHRF1和泛乙酰化赖氨酸抗体进行Western Blot分析。在免疫沉淀(IP)中使用免疫球蛋白G(IgG)作为UHRF1抗体的对照。
(C)通过将纯化的GST-UHRF1和GST-MOF与乙酰辅酶A(acetyl-CoA)共同孵育进行体外乙酰化实验。对照实验在没有乙酰辅酶A的情况下进行。使用乙酰化赖氨酸抗体检测UHRF1的乙酰化情况,并通过GST抗体检测UHRF1和MOF蛋白。
(D)将对照组和MOF小分子干扰RNA(siRNA)转染到表达外源FLAG-Myc-UHRF1的HEK293T细胞中。使用FLAG抗体对细胞裂解物进行免疫沉淀,并使用针对FLAG和乙酰化赖氨酸的抗体进行Western Blot分析。
(E)在共转染FLAG-Myc-UHRF1和空载体或Myc-HDAC1的HEK293T细胞中,使用FLAG抗体进行免疫沉淀,随后使用针对泛乙酰化赖氨酸、UHRF1和HDAC1的抗体进行Western Blot分析。
(2)MOF在pre-RING连接区的K670位点周围乙酰化UHRF1。
通过质谱分析和突变实验,确定了MOF主要乙酰化UHRF1的K670位点,而K667和K668在K670突变时也可以被乙酰化。
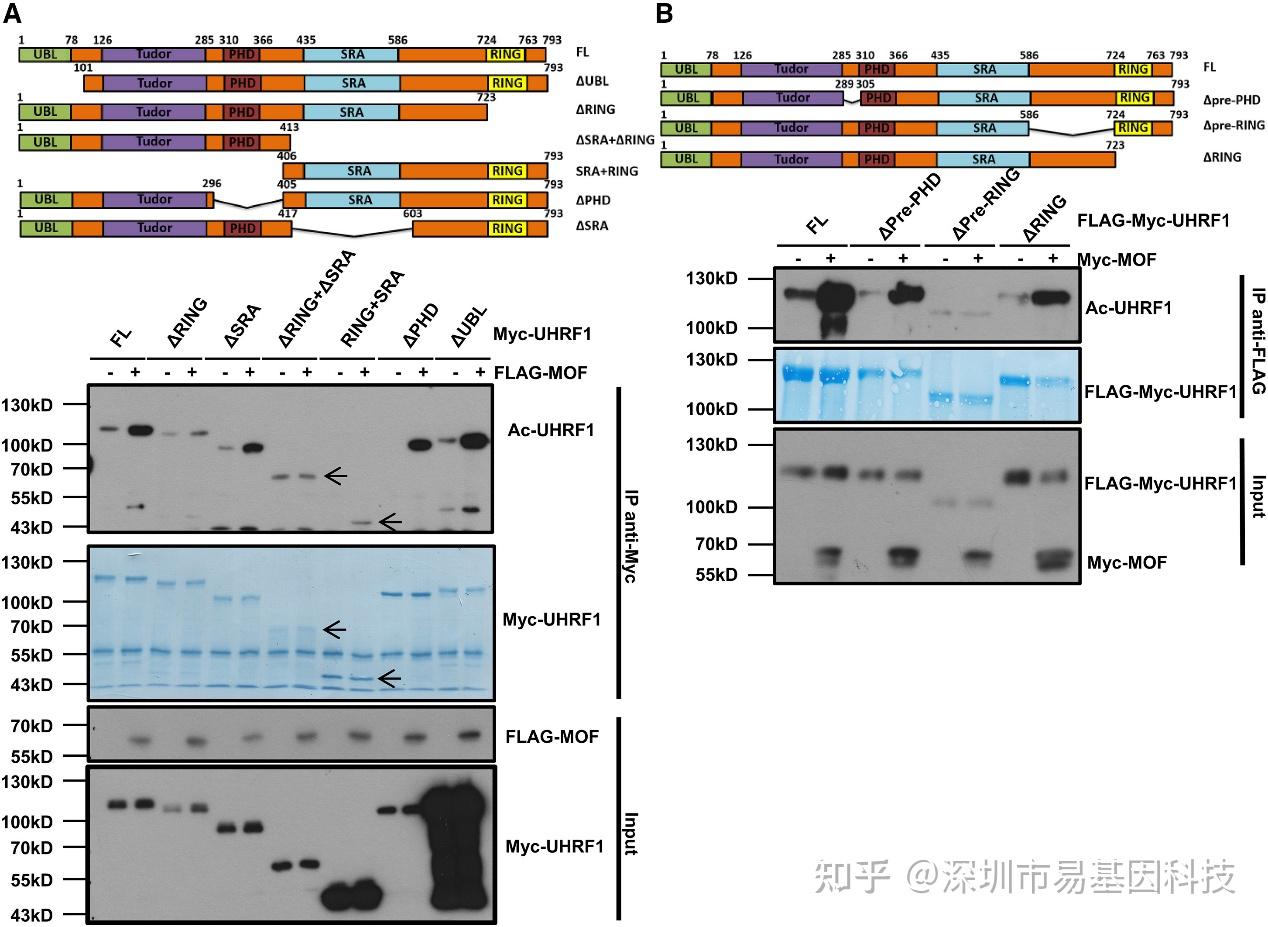
图2:MOF在pre-RING连接区乙酰化UHRF1
(A)上图展示了用于乙酰化分析的野生型UHRF1和缺失不同结构域的UHRF1突变体示意图。将不同的Myc标记的UHRF1构建体单独或与FLAG-MOF共转染到HEK293T细胞中。使用Myc抗体进行免疫沉淀(IP)后,通过Western Blot分析检测input样本和沉淀样本中的Myc、FLAG或乙酰化赖氨酸。
(B)上图展示了用于乙酰化分析的UHRF1和缺失连接区的构建体示意图。通过定点突变技术生成缺失连接区的UHRF1突变体构建体。将UHRF1构建体单独或与Myc-MOF共转染到HEK293T细胞中。使用FLAG抗体进行免疫沉淀后,通过Western Blot分析检测input样本和沉淀样本中的Myc和泛乙酰化赖氨酸。
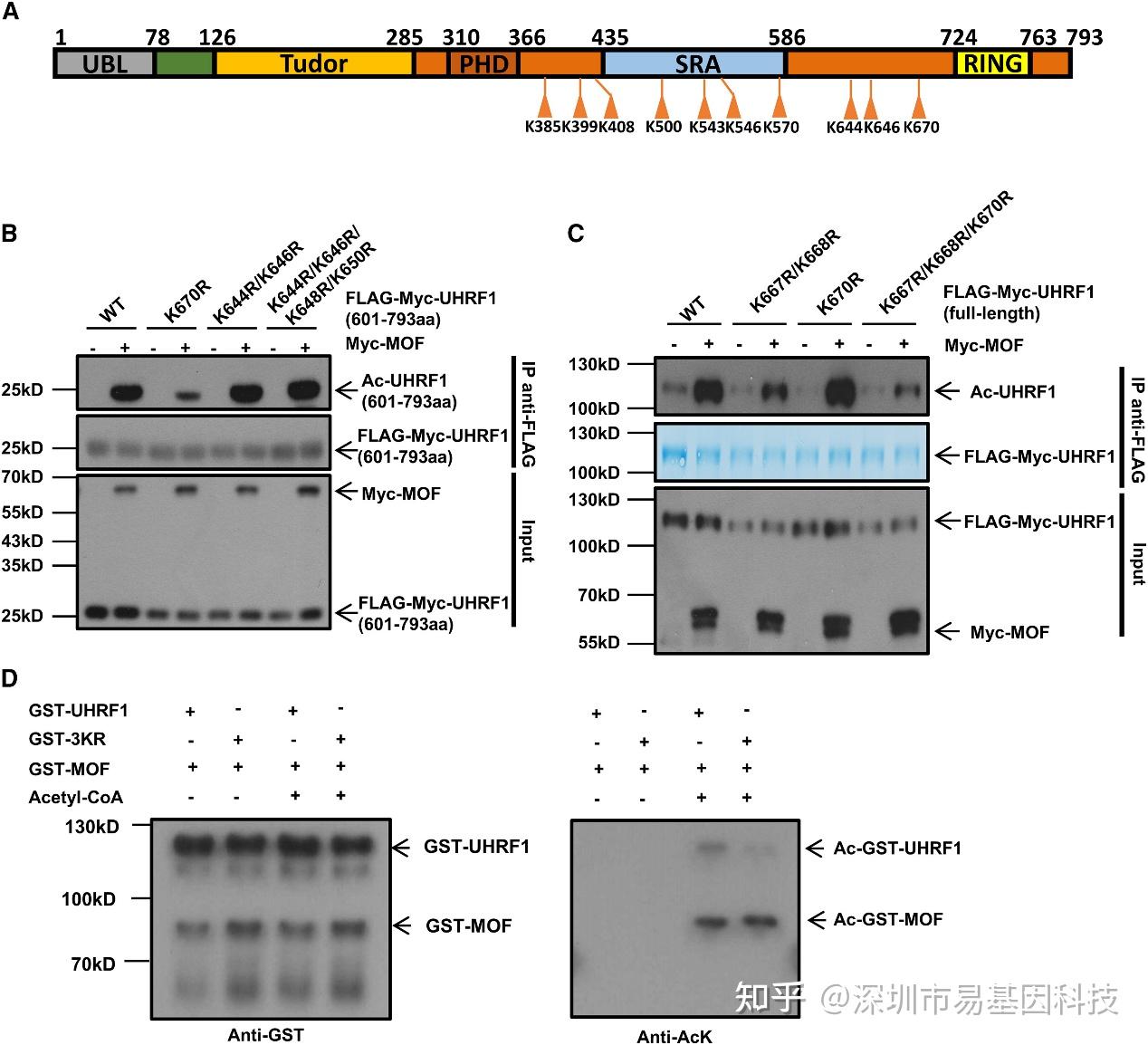
图3:通过定点突变鉴定MOF介导的UHRF1乙酰化位点
(A)通过质谱分析鉴定出的UHRF1中得分最高的10个乙酰化赖氨酸位点示意图。
(B)在有无MOF存在的情况下,野生型UHRF1片段(601-793氨基酸)及各种突变型UHRF1片段(601-793氨基酸)的乙酰化情况。外源性野生型UHRF1片段和不同突变型UHRF1片段经FLAG抗体免疫沉淀后,使用泛乙酰化赖氨酸抗体进行Western Blot分析。input样本用Myc抗体分析外源性UHRF1片段和MOF的表达情况。
(C)在FLAG-Myc-UHRF1质粒中,靶向K667/K668/K670赖氨酸簇进行了赖氨酸到精氨酸的突变。将野生型或UHRF1点突变构建体(均带有FLAG-Myc标签)单独或与Myc-MOF共转染到HEK293T细胞中。使用FLAG抗体进行免疫沉淀后,通过Western Blot分析检测Myc和乙酰化赖氨酸。
(D)通过将纯化的GST-UHRF1或突变型K667R/K668R/K670R(3KR)-UHRF1和GST-MOF与乙酰辅酶A(acetyl-CoA)共同孵育进行体外乙酰化实验。对照实验在没有乙酰辅酶A的情况下进行。使用乙酰化赖氨酸抗体检测UHRF1的乙酰化情况,并通过GST抗体检测UHRF1和MOF蛋白。
(3)MOF在G1期和S期乙酰化UHRF1,并促进DNA甲基化。
UHRF1乙酰化水平在细胞周期的G1/S期达到峰值,与UHRF1招募DNMT1的功能相一致。在UHRF1的K670、K667和K668位点突变的细胞中,DNA甲基化水平显著降低,表明这些位点的乙酰化对DNA甲基化维持至关重要。
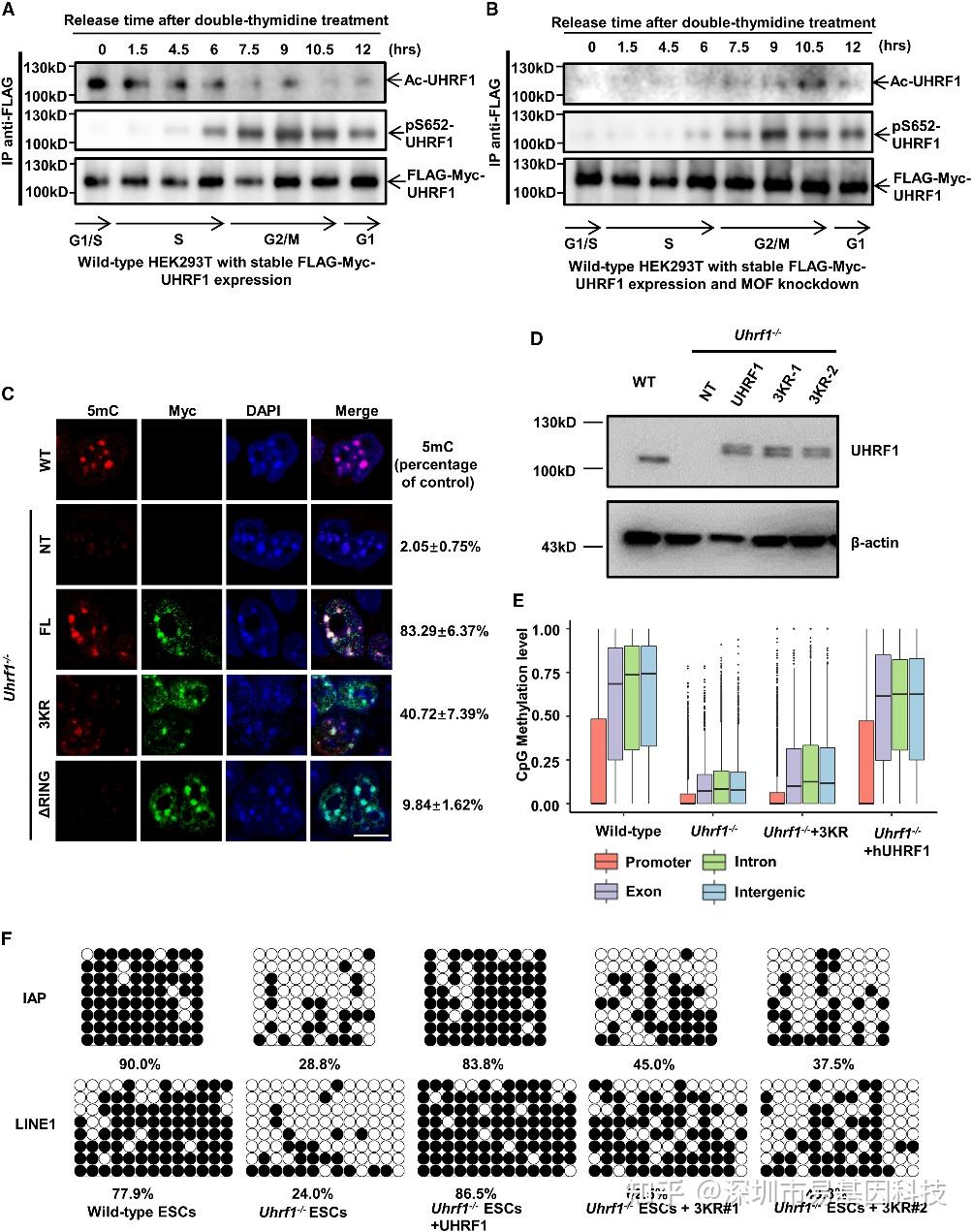
图4:UHRF1的细胞周期依赖性乙酰化及其乙酰化突变体对DNA甲基化维持的影响
(A)在稳定表达FLAG-Myc-UHRF1的HEK293T细胞中,UHRF1乙酰化水平在细胞周期中的代表性水平。在细胞周期释放后的不同时间点收集细胞裂解物,并使用FLAG抗体对FLAG标记的UHRF1进行免疫沉淀。使用乙酰化赖氨酸和pS652-UHRF1抗体分析免疫沉淀样本中UHRF1的总乙酰化和pS652磷酸化水平。使用FLAG抗体检测沉淀物中UHRF1的加载量。pS652-UHRF1水平用作细胞周期标记,因为它在G2/M期达到峰值。
(B)稳定转染野生型FLAG-Myc-UHRF1并瞬时转染MOF siRNA的HEK293T细胞通过双重胸苷阻断同步化,随后进行细胞周期释放,并进行与(A)相同的分析。
(C)在野生型和Uhrf1-/-小鼠胚胎干细胞(ESCs)中,以及通过FLAG-Myc标记的野生型UHRF1(FL)或3KR或ΔRING突变体UHRF1构建体遗传互补的Uhrf1 Uhrf1-/-小鼠胚胎干细胞(ESCs)中,使用特异性5mC抗体进行代表性免疫荧光分析。
(D)在Uhrf1 Uhrf1-/-小鼠胚胎干细胞(ESCs)中稳定表达的FLAG-Myc标记的人类野生型UHRF1和3KR突变体UHRF1通过西方印迹分析,使用UHRF1抗体进行分析。β-肌动蛋白用作加载对照。选择两个突变克隆和一个野生型克隆,其UHRF1表达水平与野生型ESCs中内源性UHRF1水平相似,用于后续研究。
(E)通过RRBS测序检测的野生型和Uhrf1 Uhrf1-/-小鼠胚胎干细胞(ESCs)以及通过FLAG-Myc标记的野生型UHRF1(hUHRF1)或3KR(克隆#2)UHRF1构建体遗传互补的Uhrf1-/-小鼠胚胎干细胞(ESCs)中,基因区域各个部分的全局CG甲基化水平。
(F)在对照ESCs、Uhrf1 Uhrf1-/-ESCs以及稳定表达人类野生型或3KR突变体UHRF1的Uhrf1 Uhrf1-/-ESCs中,通过亚硫酸盐测序分析IAP和LINE1位点的DNA甲基化状态。
(4)MOF/HDAC1介导的UHRF1乙酰化对于通过H3泛素化在DNA甲基化维持过程中高效招募DNMT1至关重要
乙酰化突变的UHRF1不能有效地招募DNMT1到异染色质区域,导致DNA甲基化缺陷。且乙酰化突变的UHRF1显著降低了其对组蛋白H3的E3泛素连接酶活性,进而影响DNMT1招募。
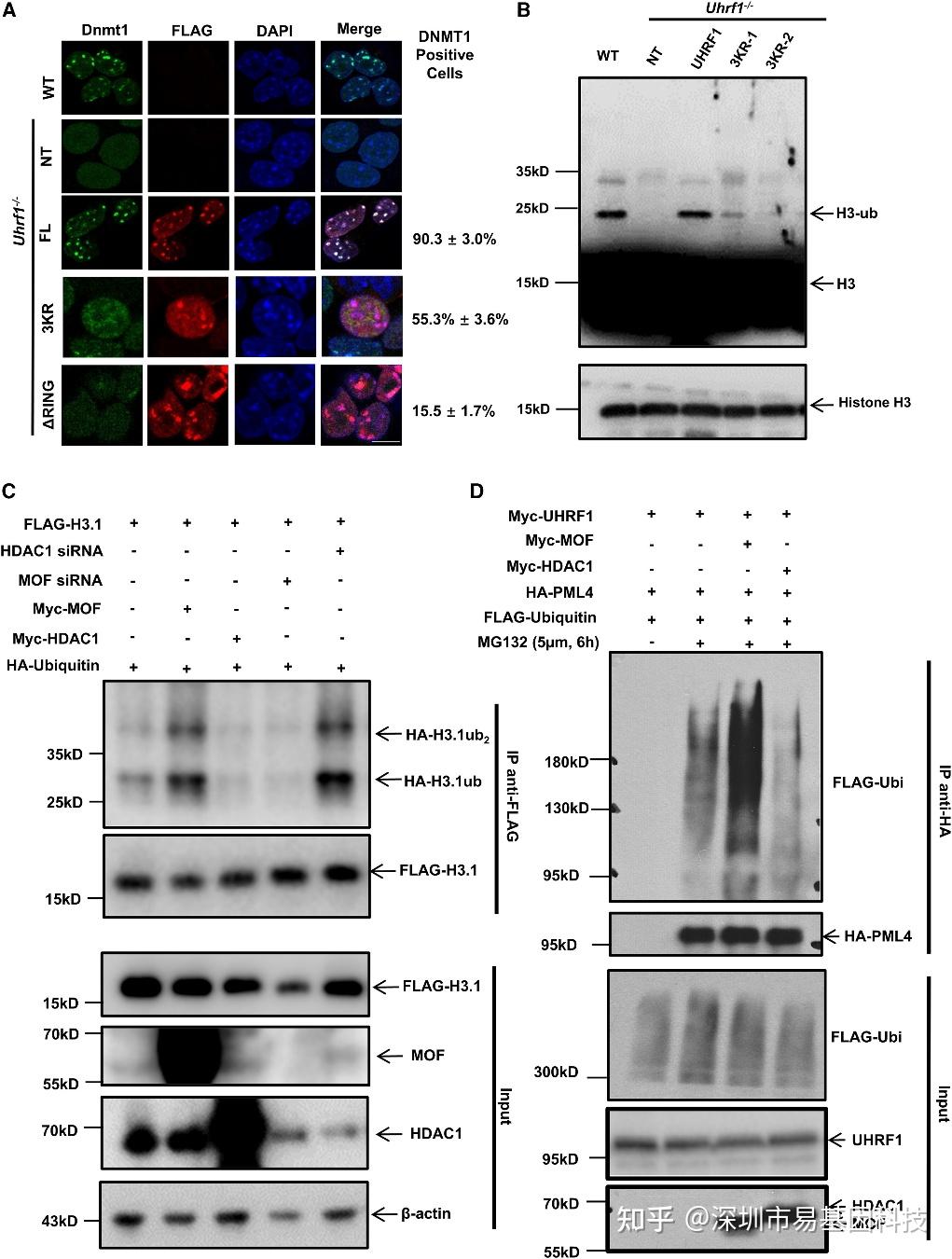
图5:UHRF1乙酰化为组蛋白H3泛素化和DNMT1招募所必需
(A)在野生型Uhrf1胚胎干细胞(ESCs)、Uhrf1-/-小鼠胚胎干细胞(ESCs)以及表达FLAG-Myc标记的野生型UHRF1或3KR突变体UHRF1或ΔRING突变体UHRF1的Uhrf1-/-小鼠胚胎干细胞(ESCs)中,对Dnmt1进行免疫荧光分析。
(B)来自至少三次重复实验的代表性Western Blot分析,用于检测野生型小鼠胚胎干细胞(ESCs)、Uhrf1-/-小鼠胚胎干细胞(ESCs)以及表达外源性人类FLAG-Myc标记的野生型UHRF1或3KR(两个克隆)突变体UHRF1的Uhrf1-/-小鼠胚胎干细胞(ESCs)中的泛素化组蛋白H3。
(C)在HEK293T细胞中组蛋白H3.1泛素化。在存在MOF、HDAC1、MOF siRNA或HDAC1 siRNA的情况下,从HEK293T细胞中免疫沉淀的外源性FLAG-H3.1的泛素化通过Western Blot进行检测。将裂解液进行FLAG抗体的免疫沉淀(IP),以沉淀外源性组蛋白H3.1,随后使用FLAG和血凝素(HA)抗体进行免疫印迹分析,以检测H3.1和HA标记的泛素。在FLAG IP样本中显示的HA标记的泛素反映了FLAG-H3.1的泛素化水平。
(D)在HEK293T细胞中PML4的泛素化。将HA-PML4、Myc-UHRF1和FLAG-泛素构建体转染到HEK293T细胞中,在有无MOF或HDAC1存在的情况下进行实验。在收获前6小时用5μM MG132处理细胞。将细胞裂解液进行HA抗体的免疫沉淀,以沉淀外源性表达的野生型HA-PML4,随后使用HA、FLAG抗体进行免疫印迹分析,以检测PML4和FLAG标记的泛素。
(5)在小鼠胚胎干细胞(ESCs)中敲低Mof会降低DNA甲基化水平。
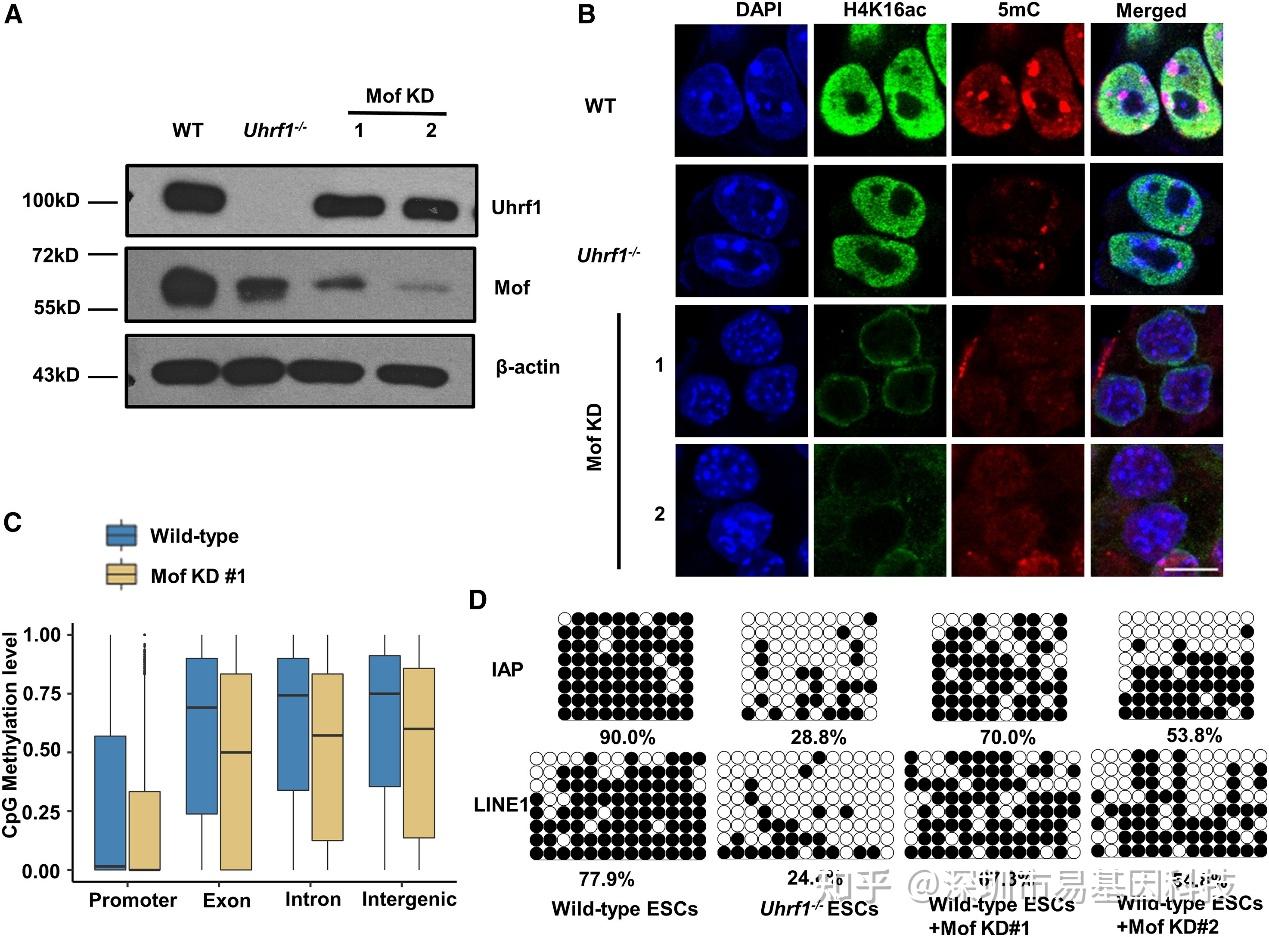
图6:在小鼠胚胎干细胞(ESCs)中敲低Mof会降低DNA甲基化
(A)通过稳定表达两种不同的KAT8短发夹RNA(shRNA),生成了两条Mof(KAT8)敲低(KD)小鼠胚胎干细胞(ESCs)系。对野生型、Uhrf1-/-和Mof敲低小鼠胚胎干细胞(ESCs)的细胞裂解液分别用MOF和UHRF1抗体进行免疫印迹分析。
(B)对野生型、Uhrf1-/-和Mof敲低小鼠胚胎干细胞(ESCs)中的5mC水平进行免疫荧光染色。对内源性H4K16ac进行染色以反映Mof乙酰转移酶活性。
(C)通过RRBS测序检测的野生型和Mof敲低(clone 1)小鼠胚胎干细胞(ESCs)基因组DNA在不同基因组区域的整体CpG DNA甲基化谱。
(D)在野生型、Uhrf1-/-和稳定敲低Mof的野生型小鼠胚胎干细胞(ESCs)中,通过亚硫酸盐测序分析IAP和LINE1位点的DNA甲基化情况。
易小结
本研究揭示了MOF对UHRF1的乙酰化修饰是DNA甲基化维持的关键调控机制。UHRF1的K670、K667和K668位点的乙酰化增强了其E3泛素连接酶活性,促进了组蛋白H3泛素化,从而有助于DNMT1招募和DNA甲基化维持。这一发现不仅为理解DNA甲基化维持的分子机制提供了新视角,还可能为相关疾病的治疗提供潜在靶点。
RRBS在本研究中的作用
RRBS(Reduced Representation Bisulfite Sequencing)是一种用于分析基因组中DNA甲基化模式的技术。在本研究中,RRBS被用于分析不同基因型小鼠胚胎干细胞(包括野生型、UHRF1敲除型和表达突变UHRF1的细胞)的全基因组DNA甲基化水平。
- 单碱基分辨率检测不同基因组区域的DNA甲基化水平,揭示UHRF1乙酰化突变对DNA甲基化维持的整体影响。
- 鉴定在不同细胞类型或处理条件下发生显著甲基化变化的基因组区域(差异甲基化区域),为理解UHRF1在DNA甲基化维持中的作用提供了基因组学证据。
- 结合基因表达数据,分析DNA甲基化变化对基因表达的潜在影响,进一步揭示UHRF1乙酰化在表观遗传调控中的功能。
关于易基因简化基因组甲基化测序(RRBS)研究解决方案
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。
为适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力适用低起始量DNA样本(5ng)量多维度甲基化分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向基因组测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和微量样本复用检测,使其具有高度可扩展性,并适用于有限的样本和单个细胞基因组CG位点覆盖高达15M以上。
技术优势:
- 起始量:100ng gDNA;
- 单碱基分辨率;
- 多样本的覆盖区域重复性可达到85%-95%、测序区域针对高CpG调控区域,数据利用率更高;
- 针对性强,成本较低;
- 基因组CG位点覆盖高达10-15M,显著优于850K芯片。
应用方向:
RRBS/dRRBS/XRBS广泛应用于动物,要求全基因组扫描(覆盖关键调控位点)的:
- 队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选
- 复杂疾病及肿瘤发病机制等甲基化研究
- 模式动物发育和疾病甲基化研究
| 技术参数 | RRBS | DRRBS | cfDNA-RBS | Micro-RBS | sc-RBS |
|---|---|---|---|---|---|
| 原理 | MspI酶切+连接接头 | 多酶切+连接接头 | CCGG邻近片段连接接头 | CCGG邻近片段连接接头 | CCGG邻近片段连接接头 |
| 样本要求 | 1μg | 1μg | 1ng | 1ng | 单细胞/1-10个细胞 |
| 测序数据量 | 10G | 15G | 20G | 20G | 2G |
| 5XCG位点覆盖 | 6M | 8M | 6M | 10M | 4-8M |
易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化、cfDNA)和表观转录组学(m6A、m5C、m1A、m7G、ac4C、RNA与蛋白互作)、DNA与蛋白互作及染色质开放性技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900。
参考文献:
Xu X, Wang H, Han H, Yao Y, Li X, Qi J, Cai C, Zhou M, Tang Y, Pan T, Zhang Z, Yang J, Wu D, Han Y. Clinical characteristics and prognostic significance of DNA methylation regulatory gene mutations in acute myeloid leukemia. Clin Epigenetics. 2023 Mar 29;15(1):54. pii: 10.1186/s13148-023-01474-0. doi: 10.1186/s13148-023-01474-0.
相关阅读:
1. Nature | 易基因DNA甲基化测序助力人多能干细胞向胚胎全能8细胞的人工诱导
2. Cell|易基因微量DNA甲基化测序助力中国科学家成功构建胚胎干细胞嵌合体猴,登上《细胞》封面
3. 项目文章 | WGBS+RNA-seq揭示松材线虫JIII阶段形成过程中的DNA甲基化差异
4. 项目文章|WGBS揭示Vpr蛋白在HIV-1感染中对CD4+ T细胞DNA甲基化变化的作用
5.项目文章 | WGBS+RNA-seq揭示黄瓜作物的“源-库”关系受DNA甲基化调控
6. 项目文章 | WGBS等揭示SOX30甲基化在非梗阻性无精症中的表观遗传调控机制
7. 项目文章|WGBS+RNA-seq揭示PM2.5引起男性生殖障碍的DNA甲基化调控机制
8.项目文章:oxBS揭示复发性膀胱癌的DNA甲基化和羟甲基化变化并鉴定预测PD-L1表达标记物
9.项目文章|PNAS:中国农大田见晖教授团队揭示DNA甲基化保护早期胚胎线粒体基因组稳定性
10.年终盘点 | 易基因2023年度DNA甲基化研究项目文章精选
11. 项目文章:泪腺RRBS+RNA-seq揭示Sjögren综合征相关干眼症的潜在基因
12. 项目文章 | RRBS揭示基于DNA甲基化驱动基因的肾透明细胞癌预后模型的鉴定和验证
13. 项目文章|Nature子刊:RRBS等揭示IDH1-R132H突变通过DNA甲基化加剧顺铂诱导肾毒性
14.项目文章|基于dRRBS技术的精液DNA甲基化年龄预测:全基因组标记鉴定和模型开发
15.项目文章|RRBS揭示中药材滇重楼DNA甲基化变化及花粉调控种子休眠的表观遗传机制
16. 3文一览:简化甲基化测序(RRBS)技术优势及研究成果(医学+物种保护+农学)
17. 技术推介 | 简化基因组甲基化测序研究解决方案
18. 技术推介 | 全基因组重亚硫酸盐甲基化测序(WGBS)
来源链接:https://www.cnblogs.com/E-GENE/p/18815005









没有回复内容